
导语
为癌症患者制定适当治疗方案的第一步是确定其癌症类型,包括确定原发部位,即癌症开始的器官或身体部位。目前,恶性肿瘤的诊断往往依赖于肿瘤的组织病理学分类以及其他特征,例如突变分析和临床表现等。但肿瘤在生长过程中,肿瘤细胞经过多次分裂增殖会呈现出分子生物学或基因方面的改变,导致的肿瘤异质性可能会影响病理学家的诊断。此外,还有部分癌症的起源细胞类型目前仍不清楚。
当前,机器学习分类器已成为处理放射学和组织病理学图像的颇具前景的新工具。近日,麻省理工学院科赫综合癌症研究所和麻省总医院的研究团队合作在Cancer Discovery发表了题为“Developmental Deconvolution for Classification of Cancer Origin”的文章。研究团队开发了一种新型深度学习模型“D-MLP”,可通过分析、比较早期细胞发育和分化相关的基因表达数据对未知原发性癌症(CUP)进行分类。该研究提供了肿瘤发育起源的系统图谱,为病理诊断提供了有效的工具,并提示发育分类可能是助力患者肿瘤治疗有潜力的方法。

为了应对机器学习分类目前存在的诸多挑战,研究团队使用了两个综合图谱数据:癌症基因组图谱(TCGA)和小鼠器官发生细胞图谱(MOCA)。TCGA包含33种肿瘤和正常组织类型的表达数据,并附有诊断信息;MOCA包含单细胞表达谱(scRNAseq),剖析了小鼠受精后第 9.5天到第13.5天(E9.5-E13.5)器官发生的全过程(对应于人类的E22-E44),并带有发育轨迹信息。MOCA代表了最完整的哺乳动物器官发生图谱,并且MOCA发育谱系与人类直系同源谱系显示出高度相似性。
研究团队系统地比较了上述两个图谱数据,并分析了其在构建诊断工具中的作用,最终构建了人类肿瘤发育图谱。
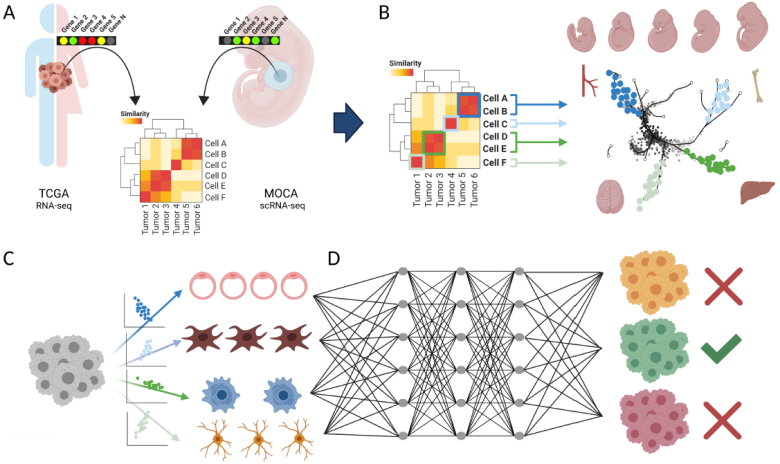
图1. 研究流程模式图。来源:Cancer Discovery
研究团队设计利用健康正常细胞之间以及不同类型癌症之间的差异建立了一个机器学习模型,可对不同癌症患者进行准确地预测、诊断。一个模型如果考虑了太多癌症基因表达的特征,其在预测新数据时反而会不准确;但通过缩小特征数量来简化模型,又可能会遗漏涉及癌症类型准确分类的重要信息。
为了在减少特征数量和提取最相关信息之间取得平衡,研究团队将模型重点放在癌细胞发育途径改变的轨迹上。将56个单细胞器官发育轨迹映射到33种癌症类型的10,000多个肿瘤转录组中,并将肿瘤转录组反卷积为个体发育轨迹的信号,对肿瘤发育轨迹进行了比较。研究团队观察到了具有强正相关和负相关的发育信息轨迹、与肿瘤具有混合关系的轨迹以及许多其他关联。
图2. 发育反卷积捕获正常和恶性细胞的信号。来源:Cancer Discovery
接下来,研究团队将肿瘤和胚胎细胞中发育基因表达模式之间的相关性转化为机器学习模型。将来自TCGA的肿瘤样本基因表达反卷积为与发育轨迹中特定时间点相对应的单个发育成分,并为每个发育成分分配数值,基于此构建了一个机器学习模型,称为发育多层感知分类器(D-MLP),该分类器可对肿瘤发育成分进行评分,并预测其起源。
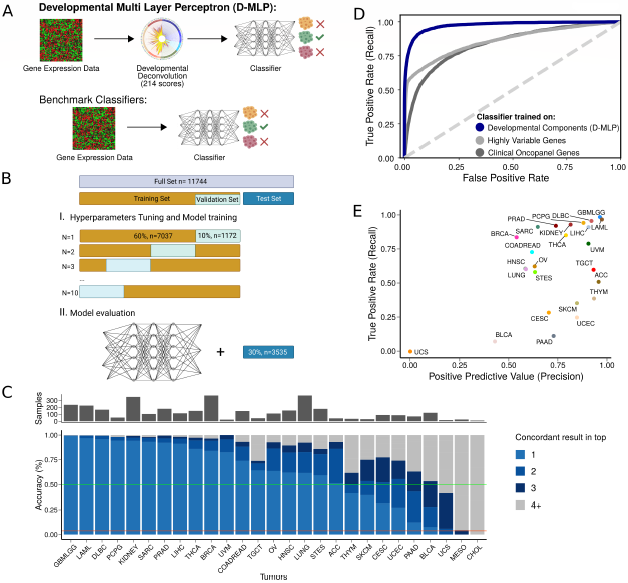
图3. D-MLP分类器的构建与检验。来源:Cancer Discovery
未知原发性癌症(CUP)是一类转移性肿瘤的统称,其占所有恶性肿瘤的3%~5%,具有侵袭性特征和较差的患者预后。由于无法确定CUP患者原发性癌灶的位置,临床医生很难通过常规诊断方法对患者进行诊断、治疗,目前亟需新的方法来解决CUP患者的诊断困境。
研究团队对D-MLP分类器进行了训练,并将其应用在52个CUP患者的肿瘤样本中。结果显示,D-MLP分类器准确将这些肿瘤分为四类,并生成了指导这些患者诊断和治疗的预测和其他信息。
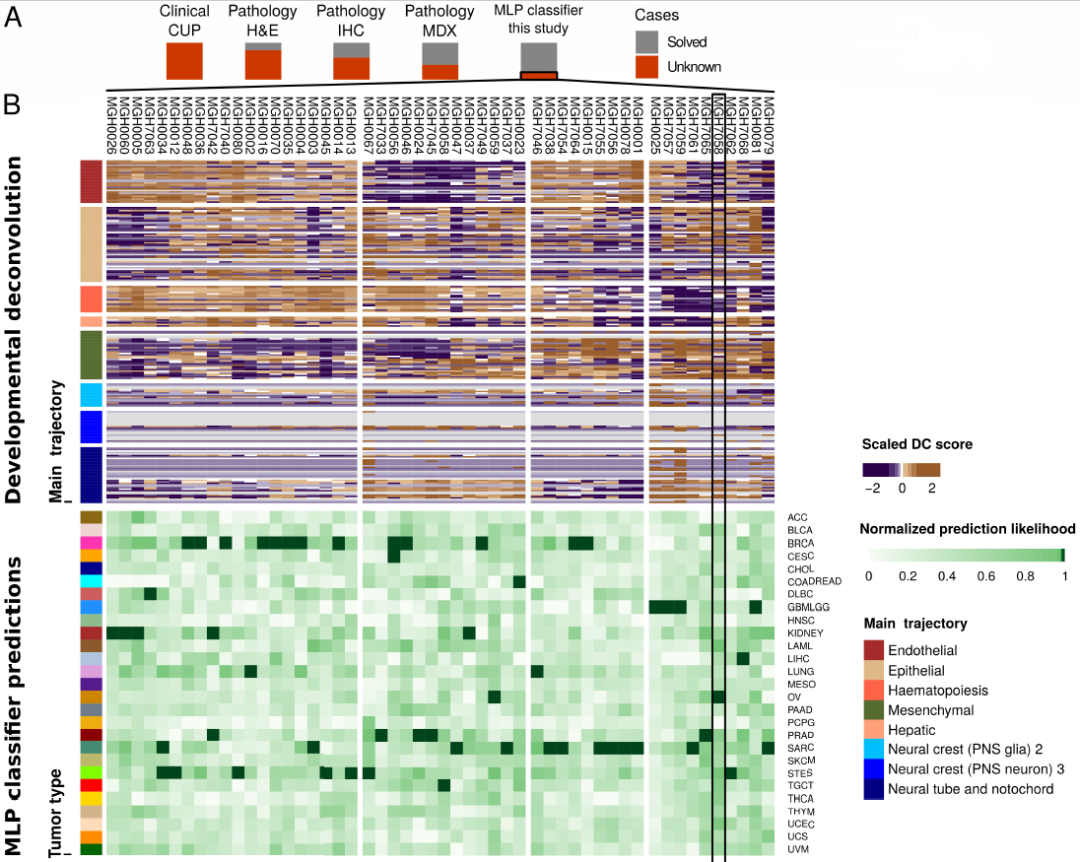
图4. D-MLP分类器对CUP患者的诊断。来源:Cancer Discovery
研究团队在一位具有乳腺癌病史的患者中验证了D-MLP模型的预测性能。该患者腹部周围的液体空间中显示出侵袭性癌症的迹象,但肿瘤学家无法找到肿瘤块,也无法使用现有工具对癌细胞进行分类。D-MLP强烈预测了该患者卵巢癌的发生。在患者首次就诊六个月后,经过广泛的额外临床检查,医生的确在患者的卵巢中发现了一个肿块,并证实其是肿瘤的起源。
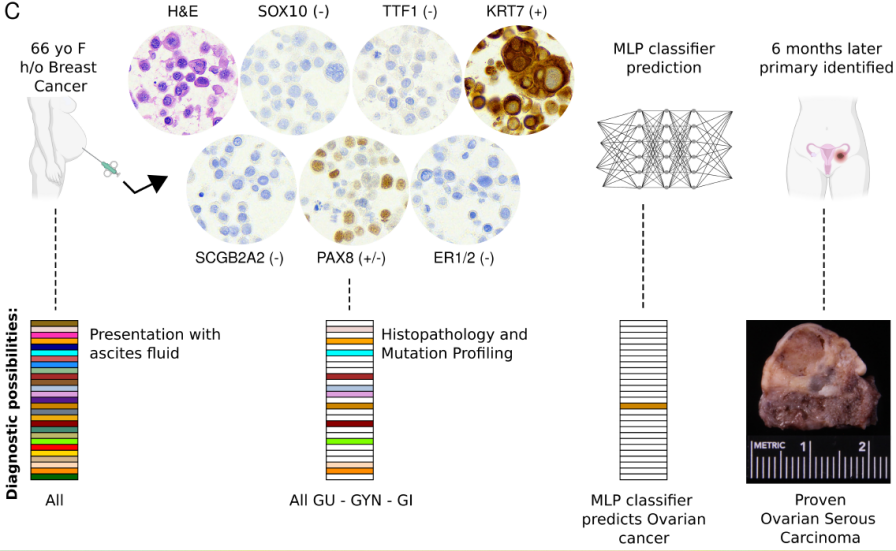
图5. D-MLP分类器对CUP患者的诊断-案例验证。来源:Cancer Discovery
综上所述,研究团队分析比较了TCGA肿瘤样本和MOCA单细胞数据,构建了人类肿瘤发育图谱,系统地量化了癌症生物学和发育程序之间的相似性。使用该图谱构建了一个能高精度预测肿瘤类型的D-MLP分类器。该研究证明可通过发育轨迹和肿瘤特征的整合对其他未分类的肿瘤实体进行诊断。
文章第一作者麻省理工学院博士后Enrico Moiso表示:“单细胞解析工具极大地改变了我们研究癌症生物学的方式,但我们如何使这场革命对患者产生影响是另一维度的难题。随着发育细胞图谱的出现,特别是那些关注器官发生早期阶段的图谱,如MOCA,我们可以将工具扩展到组织学和基因组信息之外,为分析、识别肿瘤以及开发新疗法提供了思路。”
参考文献:
1. Enrico Moiso et al, Developmental Deconvolution for Classification of Cancer Origin, Cancer Discovery 2022.
2. Lu MY, et al. AI-based pathology predicts origins for cancers of unknown primary. Nature 2021.
3. Cao J, et al. The single-cell transcriptional landscape of mammalian organogenesis. Nature 2019.
声明:本文来源测序中国,仅为交流学习。内容仅代表作者个人观点,望大家理性判断及应用。














